
L’hybridation entre espèces, associée à la duplication de génome (on parle d’allopolyploïdie), est un processus d’évolution et de spéciation majeur chez les plantes. Lors de sa thèse de doctorat dirigée par Karine Alix et Johann Joets dans l’équipe GEvAD, Paulina Martinez Palacios et coll. ont utilisé des colzas néo-synthétisés allotétraploïdes pour démontrer le rôle important des petits ARN non codants comme agents de contrôle de la stabilisation du nouveau génome allopolyploïde. Ces résultats ont été publiés dans la revue “Molecular Biology and Evolution”.
Fig 1 : culture expérimentale de Colza
Notre étude a eu pour objectif de caractériser la réponse globale de ces petits ARN non codants à un événement d’allopolyploïdie chez les Brassica. Le colza (Brassica napus) est une espèce allotétraploïde qui trouve son origine dans l’hybridation spontanée entre un navet (B. rapa) et un chou (B. oleracea). Nous avons caractérisé les niveaux d’accumulation des populations de petits ARN chez trois colzas néo-synthétisés, issus de croisements contrôlés entre un chou et un navet réalisés au laboratoire à Rennes, en comparaison de leurs progéniteurs diploïdes.
En utilisant les techniques récentes de séquençage haut-débit, les analyses de près de 20 millions de séquences ont permis de caractériser la dynamique générale des populations de petits ARN non codants. Nous avons démontré qu’ils répondaient de manière immédiate et transitoire à l’événement d’allopolyploïdie, et ce de manière identique chez les trois colzas néo-synthétisés étudiés. Cette réaction « dans l’urgence » d’augmentation des populations de petits ARN de 21nt à action PTGS correspond essentiellement à des petits ARN ciblant des éléments transposables ou des séquences endogènes de type viral. Au fil des générations, des petits ARN ciblant les mêmes séquences mais de 24nt à action TGS apparaissent et permettraient un contrôle à plus long terme de ces séquences, dont l’activité est souvent source d’instabilité génomique. Un système « ceinture et bretelles », en quelque sorte…
Figure 2, adaptée de l’article qui est publié en open access CC BY-NC. La dynamique globale des petits ARN interférents (siRNAs) en réponse à l’allopolyploïdie se traduirait par un passage progressif du mode de régulation PTGS contrôlé par des petits ARN de 21nt (système de contrôle d’urgence) au mode de régulation TGS contrôlé par des petits ARN de 24nt (système de contrôle à plus long terme). La figure résume uniquement les résultats obtenus lors de notre étude. La zone orange correspond à l’étape d’hybridation F1, moment de la rencontre des génomes différenciés où a lieu l’étape du Genome Shock selon B. McClintock, et du premier colza néo-synthétisé S0. La portion en gris de la figure reste hypothétique car elle n’a pas été analysée durant cette étude.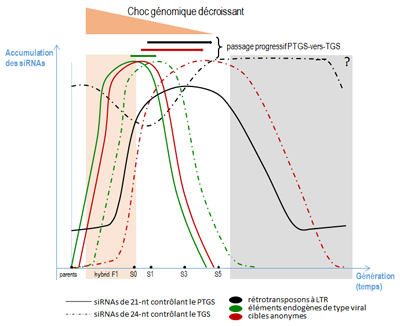
Nos résultats montrent la mise en place d’une réorganisation immédiate de la production des petits ARN en réponse à l’allopolyploïdie, permettant un contrôle efficace de la réactivation transcriptionnelle de différents éléments « non-codants » du génome, mais également une régulation ciblée de gènes impliqués dans des réponses aux stress. L’accumulation différentielle des petits ARN permettrait la mise en place progressive d’une certaine stabilité génomique nécessaire aux premières étapes de formation d’un néo-allopolyploïde. Cela nous amène à penser que sans ces populations de petits ARN, mobilisés comme des « gardiens du génome », le nouvel événement d’allopolyploïdie n’aurait que peu de chance de réussite, laissant la part belle au choc génomique…
Référence
Contact
Karine AlixGlossaire
- Allopolyploïdie
Evénement de spéciation originaire d’une hybridation interspécifique associée à une duplication de génome complet. De nombreuses espèces cultivées sont allopolyploïdes. Par exemple, le blé tendre (Triticum aestivum, AABBDD) est le résultat de l’hybridation entre trois espèces différentes.
- Action des petits ARN non codants
De manière générale, les petits ARN ont des fonctions de régulation liées à leur taille : ceux de 21 nucléotides (nt) vont agir au niveau post-transcriptionnel (action PTGS pour post-transcriptional gene silencing) en dégradant le transcrit ciblé ou en bloquant sa traduction, alors que ceux de 24nt vont réprimer la transcription du gène ciblé (action TGS pour transcriptional gene silencing) directement au niveau de l’ADN.