Dans un article publié récemment dans la revue Genetics, Matthieu Falque et Olivier Martin présentent CNVmap, un logiciel destiné à cartographier des variants sructuraux à partir de données servant à la construction de cartes génétiques.
Une grande partie des découvertes en génétique depuis un siècle ont été rendues possibles par la construction de cartes de liaison génétique. Ces cartes permettent de déduire la position d’un gène sur les chromosomes à partir de l’analyse de la ségrégation(1) de marqueurs génétiques dans une population d’individus descendant de parents connus. Il est ainsi possible d’identifier une position sur la séquence d’ADN, dont la variation est responsable des différences observées entre individus pour un caractère, par exemple d’intérêt médical (prédisposition à une maladie) ou agricole (tolérance aux stress, qualité, productivité). Ces différences de séquence d’ADN sont diverses mais les plus étudiées sont les substitutions de bases (appelées Single-Nucleotide Polymorphisms ou SNP) ou de petites insertions-délétions de quelques bases.
Cependant, il existe d’autres types de variation de séquence d’ADN, qui impliquent des régions beaucoup plus grandes (supérieures à 1 000 bases), que l’on appelle des variants structuraux (SV). Il peut s’agir par exemple de régions présentes en un nombre différent de copies selon les individus (Copy-Number Variant ou CNV), ou de régions présentes chez certains individus et pas d’autres (Presence-Absence Variant ou PAV). Ces variants structuraux peuvent également être responsables de différences entre individus pour des caractères importants et méritent à ce titre d’être identifiés. Pour cela, différentes approches existent, dont les plus utilisées sont fondées sur le séquençage de l’ADN. Hélas, les SV de grande taille sont parfois extrêmement difficiles à identifier car les technologies de séquençage produisent de nombreux fragments de séquence de taille souvent beaucoup plus petite que celle des événements recherchés.
Dans un article publié récemment dans la revue Genetics, Matthieu Falque et Olivier Martin, de l’UMR Génétique Quantitative et Évolution - Le Moulon, présentent une méthode originale et un logiciel nommé CNVmap (logiciel libre sous forme d’un paquet R disponible à tous) qu’ils ont développés pour détecter et cartographier des variants structuraux à partir des données servant à la construction de cartes génétiques. De très nombreux jeux de données de ce type ont été produits ces dernières années, en particulier dans le domaine agronomique, surtout depuis que des méthodes de génotypage à haut-débit permettent d’obtenir des cartes extrêmement précises.
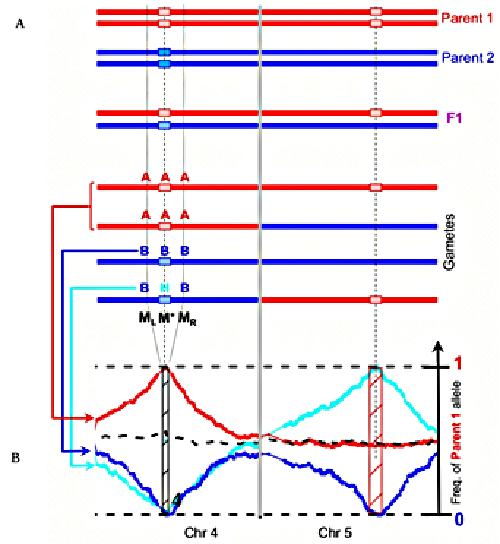
Figure : Schéma de méthode de détection
Illustration de la méthode permettant de détecter une région dont le parent 1 possède une deuxième copie, absente chez le parent 2. Ces deux parents avaient été croisés pour donner un hybride F1, à partir duquel avait été produite une population de gamètes, ou de lignées dérivées de ces gamètes. Chaque individu de cette population avait été analysé avec des marqueurs pour identifier l’allèle (variant) du parent d’origine, dans le but de construire une carte génétique. L’analyse de ces données à l’aide du logiciel CNVmap permet de détecter et de localiser sous forme de pics le long des chromosomes la deuxième copie (précédemment inconnue) de la région. Le logiciel détecte les marqueurs candidats à la duplication, puis génère les profils de fréquence allélique correspondants et, en analysant les pics présents sur ces profils, détecte automatiquement 33 types diférents de CNV. Chaque copie est enfin positionnée sur le chromosome qui la porte.
Ce nouveau logiciel permet de donner une deuxième vie aux données de cartographie génétique pour détecter et positionner des régions dont le nombre de copies diffère entre les parents dont est issue la population de cartographie. L’objectif du logiciel CNVmap est de permettre à tous les chercheurs produisant des données de ségrégation génétique de détecter facilement des réarrangements structuraux entre les génomes des parents de leur population grâce à une procédure entièrement automatisée.
(1) Le terme de ségrégation génétique désigne la façon dont les gènes d’un individu se répartissent dans les gamètes produits lors de la méiose, et donc dans la descendance de cet individu.
Référence
Contact : Matthieu Falque