
Dans un article publié dans Genome Research, des chercheurs du laboratoire GQE-Le Moulon, en collaboration avec des collègues du LEPSE à Montpellier, ont étudié les bases génétiques et moléculaires de la réponse à la sécheresse chez le maïs en associant des variations observées à l’échelle du génome à celles observées au niveau des protéines et des caractères phénotypiques.
Dispositif expérimental au LEPSE
L’expérience originale a consisté à caractériser des mêmes plantes à trois niveaux différents : le génome à l’aide de marqueurs SNPs, un ensemble de caractères physiologiques connus pour être affectés par le déficit hydrique, et des phénotypes moléculaires intermédiaires : l’abondance des protéines présentes dans les feuilles, ou protéome.
Un panel de 254 génotypes de maïs, représentatifs de la diversité génétique du groupe des maïs dentés, a été cultivé en serre sous deux conditions : une condition normale d’irrigation et une condition de déficit hydrique. Les données massives qui ont été collectées (1 million de marqueurs SNP, mesure d’une dizaine de caractères phénotypiques et quantification de 2000 protéines sur 1000 plantes) ont permis de réaliser une étude de génétique d’association à l’échelle du génome pour les caractères phénotypiques et pour l’expression des protéines dans chacune des conditions de culture, et aussi d’analyser les corrélations entre protéines et entre protéines et caractères phénotypiques.
Figure 1 : Des modules de protéines co-exprimées ont été établis (à droite), permettant la détection de QTL de co-expression. À gauche, exemple de co-localisation entre QTL d’abondance de protéines (en rose) de caratères écophysiologiques de la plante (en vert) et de modules de protéines co-exprimées (en bleu). Les lignes bleues correspondent à des co-localisation sur le même SNP. ZmGh3 est le gène candidat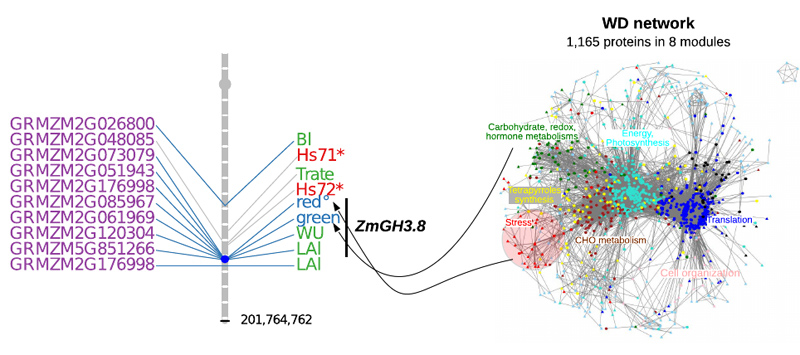
L’analyse conjointe des données génomiques, phénotypiques et protéomiques montre qu’un déficit hydrique, même léger, provoque une reprogrammation du contrôle génétique des abondances de protéines et entraîne un profond remodelage du protéome. Il entraîne aussi une restructuration importante du réseau de co-expression des protéines. L’étude a permis d’identifier des régions du génome associées à la fois à des variations observées à l’échelle du protéome et à des variations observées à l’échelle de la plante entière. Ceci suggère que les variations génétiques de l’abondance de certaines protéines ou de groupes de protéines co-exprimées pourraient être impliquées dans la variation des caractères physiologiques.
Cette étude ouvre des perspectives pour l’analyse de la relation génotype-phénotype et pour la découverte de gènes d’intérêt pour l’amélioration de la tolérance à la sécheresse chez le maïs.
Contacts : Mélisande Blein-Nicolas et Michel Zivy
Référence :