L’Agence Nationale de la Recherche finance le projet EvolRec : “Démêler les causes et conséquences évolutives des variations quantitatives de la recombinaison méiotique” porté par Matthieu Falque (équipe BASE du Moulon).
Le projet met en oeuvre des approches d’évolution expérimentale chez la levure pour analyser le déterminisme génétique du nombre et de la position des crossing-overs, ainsi que le rôle de la recombinaison dans la dynamique de l’adaptation au stress.
La méiose est une division cellulaire particulière permettant la réduction d’un facteur 2 de la quantité de matériel génétique de la cellule. Elle permet de produire des gamètes haploïdes à partir de cellules
euploïdes
(par exemple diploïde). Lors de la reproduction sexuée, ces gamètes pourront ensuite avec la fécondation donner une nouvelle cellule euploïde. Au début de la méiose se produisent des échanges réciproques de matériel génétique (crossing-overs) entre chromosomes homologues par le phénomène de la recombinaison méiotique. Ce processus a deux conséquences : (1) il assure un lien mécanique entre les homologues permettant une segrégation équilibrée des chromosomes indispensable pour obtenir des gamètes viables, et (2) il permet la création dans les gamètes de nouvelles combinaisons génétiques à partir des deux chromosomes homologues du parent. La recombinaison est donc un processus fondamental pour l’évolution des génomes, la dynamique de l’adaptation des caractères et la
fitness.
Ce projet a pour thème :
- la capacité d’évolution du taux de recombinaison
Pour cela, nous utiliserons des méthodes récemment développées dans notre laboratoire pour mesurer la recombinaison à haut-débit grâce à des marqueurs fluorescents insérés sur les chromosomes, et pour trier très rapidement des millions de cellules recombinantes par ‘Fluorescence-Activated Cell Sorting’. Ces méthodes nous permettront pour la première fois de produire par sélection récurrente directionnelle des populations de levures ayant évolué pour des taux de recombinaison plus élevés ou plus faibles.
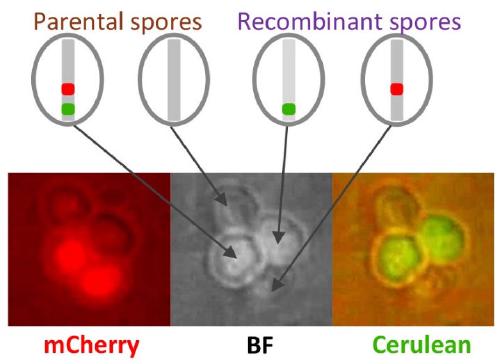
Phénotypage haut-débit du taux de recombinaison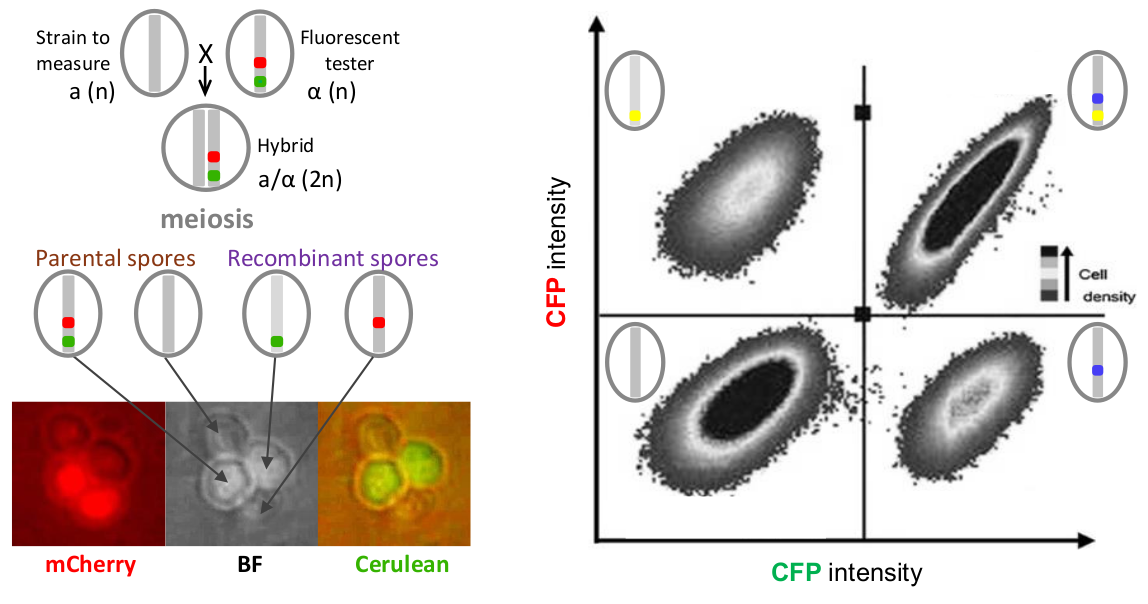
Cela permettra de mener un programme de sélection récurrente divergente sur le taux de recombinaison dans l’intervalle entre deux marqueurs à partir d’une population ‘Multiparent Advanced Generation InterCross’ développée récemment par Gianni Liti. Cycles de sélection récurrente pour augmenter le taux de recombinaison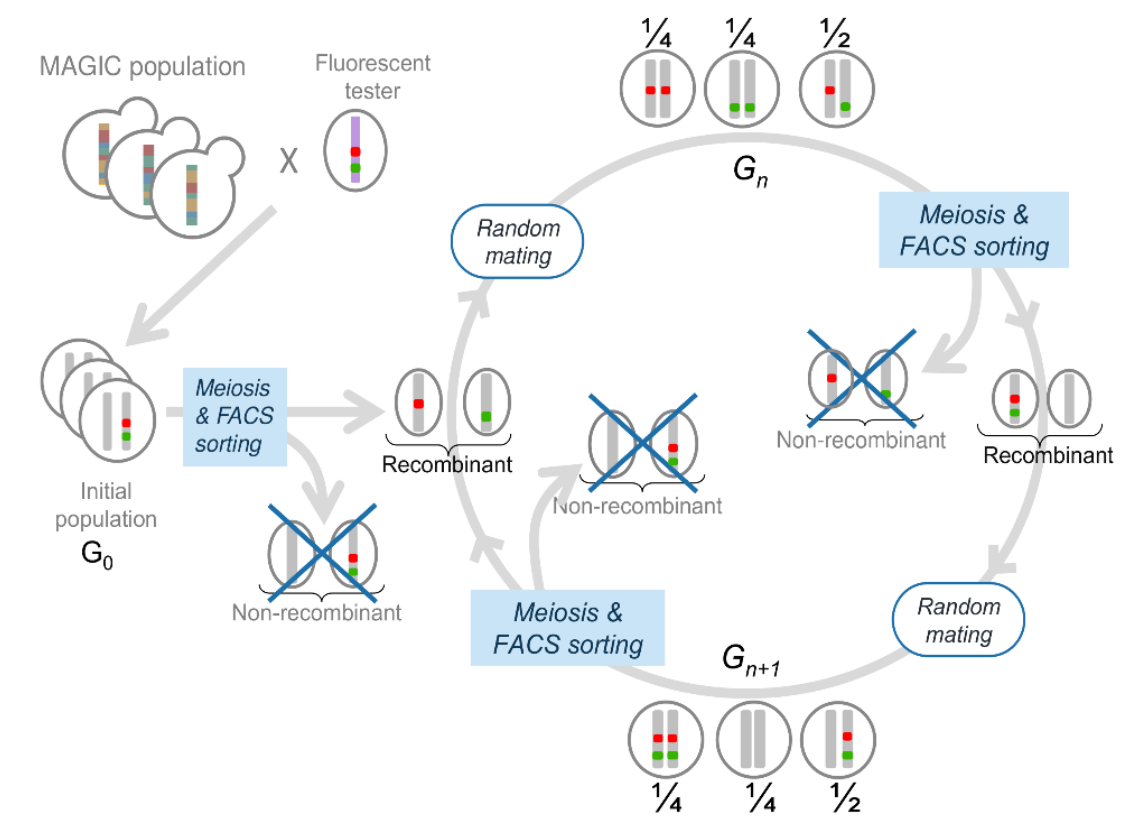
Après une dizaine de générations de sélection récurrente de ce type, on s’attend à ce que la recombinaison augmente dans l’intervalle entre les deux marqueurs ayant servi à la sélection (régulation en CIS), et/ou à ce que la recombinaison augmente plus ou moins sur l’ensemble du génome (régulation en TRANS).
- les déterminants génétiques CIS et TRANS de la variation quantitative du nombre et de la répartition des crossovers
Les populations de levure obtenues après l’expérience de sélection directionnelle pour le taux de recombinaison seront ensuite séquencées en mélange. Cetta approche de “pool-seq” permettra ainsi d’identifier les déterminants génétiques sous-jacents à la réponse à la sélection sur la recombinaison. En effet, parmi les différents allèles présents dans la population initiale, certains vont localement augmenter ou diminuer en fréquence à proximité des gènes affectés par la sélection. En étudiant les fréquences alléliques parentales le long du génome, il sera possible de localiser des pics correspondant aux signatures de sélection pour identifier les gènes impliqués.
Nous étudierons par ailleurs les profils de recombinaison génome-entier à très haute résolution par séquençage de tétrades, pour étudier l’effet des séquences agissant en CIS sur ces profils. Par exemple, nous pourrons observer si différents niveaux d’hétérozygotien, voire des variatinos structurales, peuvent modifier localement la recombinaison.
- les inter-relations entre recombinaison et adaptation au stress
L’objectif de cette partie est d’étudier expérimentalement le rôle joué par le taux de recombinaison dans la dynamique de l’adaptation au stress. Pour cela, deux types d’expériences d’évolution seront menées sur la levure S. cerevisiae, en alternant des générations de reproduction sexuée et des phases de croissance végétative sous stress salin:
- en faisant évoluer sous stress les populations issues de la sélection divergente sur le taux de recombinaison (cf 1.)
En effet, ces populations présenteront à la fois une base génétique large et des valeurs moyennes de recombinaison soit hautes, soit basses. En les faisant évoluer sous sélection (phases de croissance végétative en milieu stressant riche en sel), il devrait être possible d’étudier rôle de la recombinaison dans l’adaptation au stress, par exemple de tester si un taux de recombinaison plus élevé accélère le processus d’adaptation au stress en créant de nouvelles combinaisons haplotypiques à partir de la diversité génétique présente initialement dans la population.
- en mesurant les variations de recombinaison qui apparaissent suite à des générations de sélection sous stress salin
Pour cela, les populations initiales auront la même large base génétique que celle utilisée pour l’expérience de sélection sur le taux de recombinaison (populations MAGIC), mais n’auront pas subi de sélection directionnelle pour le taux de recombinaison. Elles évolueront sous fort stress salin, et à l’issue du processus d’adaptation, nous étudieront dans quelle mesure le taux de recombinaison aura corrélativement également évolué.
Informations complémentaires :
Partenaires du projet :
-
Gini Liti, Institut de Recherche sur le Cancer et le vieillissement (IRCAN), Université de Nice
-
Olivier Martin, Institut des Sciences des Plantes Paris-Saclay (IPS2), INRAE
Montant alloué : 463 464 €
Durée : 4 ans
Contact à GQE-Le Moulon : Matthieu Falque